Dans le processus de réalisation d'études génétiques, nous rencontrons souvent des échantillons d'ARN insuffisants, par exemple pour étudier de minuscules tumeurs buccales anatomiques, même des échantillons unicellulaires, et des échantillons de mutations génétiques spécifiques qui sont transcrites à de très faibles niveaux dans les cellules humaines.Bien sûr, pour le test COVID-19, si les écouvillons ne sont pas au bon endroit ou pas assez de fois lors du prélèvement, la taille de l'échantillon sera très faible, c'est pourquoi la Commission de la santé et de la planification familiale est sortie il y a deux jours et réussi le test, et si l'échantillonneur d'acide nucléique n'a pas prélevé six échantillons, vous pouvez le signaler.
La sensibilité du réactif est importante car nous avons tel ou tel problème, alors que pouvons-nous faire pour améliorer la sensibilité de la RT-PCR ?
Avant de discuter des solutions possibles, mentionnons deux grandes complications avec la situation que nous venons de mentionner.
Tout d'abord, nous nous inquiétons de la perte d'ARN lorsque nous n'avons que quelques populations de cellules dans notre échantillon.Si des méthodes traditionnelles de séparation et de nettoyage sont utilisées, telles que la méthode sur colonne ou la méthode de précipitation des acides nucléiques, il est fort possible que les quelques échantillons soient perdus.Une solution consiste à ajouter une molécule porteuse, telle que l'ARNt, mais même dans ce cas, il n'y a aucune garantie que notre expérience de récupération soit OK.
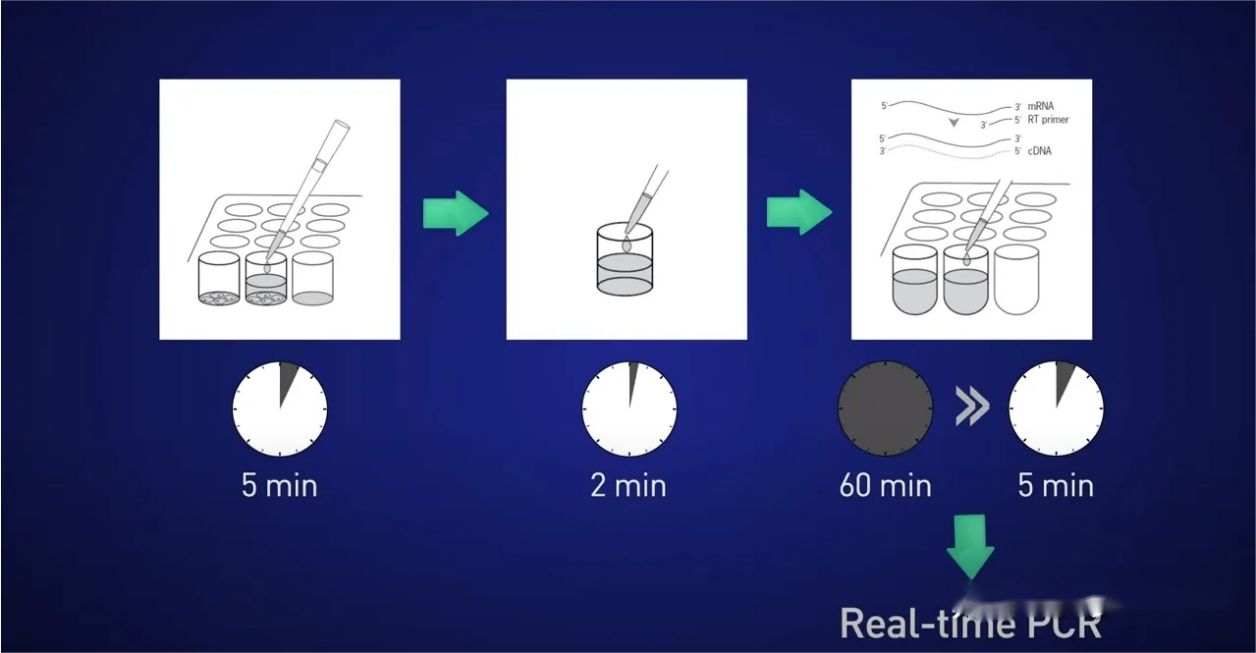
Alors, quelle est la meilleure façon ?Une bonne option pour les cellules cultivées ou les échantillons microanatomiques consiste à utiliser la lyse directe.
L'idée est de diviser les cellules pendant 5 minutes, de libérer l'ARN dans la solution, puis d'arrêter la réaction pendant 2 minutes, puis d'ajouter le lysat directement à la réaction de transcription inverse afin qu'aucun ARN ne soit perdu, et enfin de mettre directement l'ADNc résultant. dans la réaction en temps réel.
Mais que se passe-t-il si, en raison d'un point de départ limité ou d'une petite quantité d'expression du gène cible, nous pouvons recycler tout l'ARN et ne pas toujours fournir suffisamment de matrices pour obtenir un bon signal en temps réel ?
Dans ce cas, l'étape de pré-amplification peut être très utile.
Ce qui suit est un schéma pour augmenter la sensibilité après transcription inverse.Avant de commencer, il faut se demander en aval quelles cibles nous intéressent, afin de concevoir des amorces spécifiques pour ces cibles pour la pré-amplification.
Ceci peut être réalisé en créant une amorce mixte avec jusqu'à 100 paires d'amorces et un cycle de réaction de 10 à 14 fois.Par conséquent, un Master Mix spécialement conçu pour cette exigence est nécessaire pour pré-amplifier l'ADNc obtenu.
La raison de fixer le nombre de cycles entre 10 et 14 est que ce nombre limité de cycles assure un caractère aléatoire entre les différentes cibles, ce qui est crucial pour les chercheurs qui ont besoin d'informations moléculaires quantitatives.
Après la pré-amplification, nous pouvons obtenir une grande quantité d'ADNc, de sorte que la sensibilité de détection au niveau du back-end est grandement améliorée, et nous pouvons même diluer l'échantillon et effectuer plusieurs réactions PCR en temps réel pour éliminer les éventuelles erreurs aléatoires.
Heure de publication : 11 avril 2023